AI + Biología = Quantum-Si
Quantum-Si está construyendo el puente entre AI y biología molecular: de 20,000 genes a 220 millones de proteínas, una molécula a la vez.
El genoma humano contiene aproximadamente 20,000 genes. A partir de estos genes, nuestras células producen más de 220 millones de registros de secuencias proteicas distintas catalogadas en UniProt. Esta expansión exponencial desde el plano genético hasta la realidad funcional representa uno de los desafíos más profundos de la biología, y también una de las oportunidades de inversión más interesantes. Entender por qué Quantum-Si (NASDAQ: QSI) importa requiere primero comprender por qué las proteínas son fundamentalmente difíciles de analizar.
La Paradoja de Levinthal: La pesadilla Computacional de la Biología
En 1969, Cyrus Levinthal identificó lo que se convertiría en uno de los problemas más famosos de la biología computacional. Consideremos una proteína modesta de 100 aminoácidos. Cada aminoácido tiene dos ángulos en su cadena principal (phi y psi), y si cada ángulo puede adoptar solo tres conformaciones estables, la proteína enfrenta 3²⁰⁰ ≈ 10⁹⁵ conformaciones posibles. Para una proteína más realista de 150 residuos, Levinthal estimó 10³⁰⁰ conformaciones posibles.
Las matemáticas se vuelven incómodas rápidamente. Si una proteína pudiera muestrear conformaciones a 10¹³ por segundo (la escala temporal de las vibraciones moleculares), examinar todas las posibilidades requeriría aproximadamente 5 × 10³⁴ segundos, o 10²⁷ años. El universo solo tiene 10¹⁰ años de edad. Sin embargo, las proteínas se pliegan de manera confiable en milisegundos.
Esta es la paradoja de Levinthal, y muestra que las proteínas no pueden encontrar su forma correcta probando configuraciones al azar. Sería como intentar abrir una cerradura con trillones de llaves probando una por una. Los matemáticos clasifican este tipo de problemas como NP-completos, la categoría de los rompecabezas más difíciles que conocemos, aquellos donde verificar una solución es fácil pero encontrarla es exponencialmente difícil. Es el mismo tipo de complejidad que enfrentan los problemas de optimización más desafiantes en computación. En 1998, Berger y Leighton probaron formalmente que incluso versiones simplificadas del plegamiento proteico pertenecen a esta categoría intratable.
Entonces, ¿cómo lo resuelve la naturaleza? La respuesta cambió nuestra comprensión de la biología molecular. Las proteínas no exploran todas las posibilidades. En lugar de navegar un espacio plano donde cada configuración tiene igual probabilidad, siguen lo que los científicos llaman un “embudo de plegamiento”. Imagina una pelota rodando por un embudo: aunque hay baches y pequeños hoyos donde puede quedar atrapada temporalmente, la forma general del embudo la guía hacia el fondo. Ese fondo representa la estructura funcional de la proteína.
Este embudo energético tiene características medibles. La pendiente promedio es de aproximadamente -50 kcal/mol, lo que en términos prácticos significa que cada vez que una proteína forma el 10% de sus contactos correctos, libera suficiente energía para hacer el siguiente paso más probable. No es un descenso suave; hay trampas y desvíos. Pero la dirección general es clara. La evolución ha esculpido estas pendientes durante miles de millones de años, optimizando cada secuencia para que encuentre su camino eficientemente.
La física detrás de este proceso se captura en una ecuación elegante:
F(Q) = E(Q) - TS(Q)
Donde F es la energía libre (lo que determina si un estado es favorable), E es la energía de las interacciones entre átomos, T es la temperatura, y S es la entropía, una medida del desorden. Q simplemente nos dice qué tan cerca está la proteína de su forma final. La ecuación nos dice que el plegamiento balancea dos fuerzas: la tendencia a minimizar energía (E) y la tendencia natural hacia el desorden (TS). La selección natural ha afinado este balance en cada proteína, minimizando lo que los investigadores llaman “frustración”: esos molestos mínimos locales que podrían atrapar a la proteína en formas incorrectas.
Este entendimiento explica tanto el poder como los límites de herramientas modernas como AlphaFold2. El sistema de DeepMind no simula el plegamiento; aprende patrones de millones de proteínas conocidas para predecir el destino final. Es brillante en esto, logrando precisiones de 0.96 Angstroms, esencialmente a nivel atómico. Pero es como tener una fotografía del destino sin conocer el camino. AlphaFold no puede decirnos cómo una proteína viaja por su embudo energético, solo dónde termina. Y para el 30-50% de las proteínas que permanecen parcialmente desordenadas, o aquellas que cambian de forma al unirse a otras moléculas, o las que son modificadas después de ser fabricadas, incluso esa fotografía final es incompleta.
Por qué la Secuenciación Hereda el Problema del Plegamiento
Plegar proteínas como secuenciarlas enfrentan el mismo monstruo matemático, pero atacándolo desde lados opuestos. Es como la diferencia entre hornear un pastel y tratar de descifrar la receta probando el pastel terminado.
El plegamiento es el problema directo: tienes la receta (la secuencia de aminoácidos) y necesitas predecir el resultado (la estructura 3D). Con 20 tipos de aminoácidos disponibles, las combinaciones posibles crecen absurdamente rápido. Una proteína de apenas 100 aminoácidos tiene más configuraciones posibles que átomos en el universo.
La secuenciación es el problema inverso: tienes el pastel (la proteína plegada) y necesitas descifrar la receta original. Pero aquí viene el “enredo”. Las proteínas no son estáticas. Son como documentos que constantemente reciben notas al margen, tachaduras y modificaciones. Un solo gen no produce una proteína, produce docenas de versiones ligeramente diferentes.
Una forma de visualizarlo: después de que una célula fabrica una proteína, puede agregarle azúcares (glicosilación), grupos fosfato (fosforilación), o cualquiera de más de 400 tipos de modificaciones químicas conocidas. Es como si cada copia de un libro saliera de la imprenta y luego diferentes editores le agregaran notas diferentes. Algunas de estas modificaciones determinan si un medicamento funcionará o si un cáncer resistirá el tratamiento. Un solo aminoácido cambiado puede ser la diferencia entre un anticuerpo que cura y uno que no hace nada.
La espectrometría de masas, nuestra herramienta actual más poderosa, intenta resolver este rompecabezas triturando las proteínas en fragmentos pequeños y pesándolos. Funciona bien cuando ya conoces qué proteínas esperar, como buscar palabras conocidas en una sopa de letras. Pero cuando aparece algo nuevo o raro, algo que no está en tu diccionario, el método se queda corto. No puede distinguir entre leucina e isoleucina porque pesan exactamente lo mismo. No puede ver esas modificaciones sutiles que ocurren en solo algunas copias de la proteína.
El resultado es que tanto el plegamiento como la secuenciación chocan contra el mismo muro: hay demasiadas posibilidades para explorarlas todas. La naturaleza resolvió el problema del plegamiento mediante la evolución, optimizando cada proteína durante millones de años. Pero nosotros, tratando de leer y entender estas proteínas con nuestras máquinas, todavía luchamos con las limitaciones de explorar universos de posibilidades con herramientas que solo pueden ver promedios y aproximaciones.
La Restricción de la Espectrometría de Masas
Durante décadas, la espectrometría de masas ha sido el caballo de batalla para estudiar proteínas. Es como el microscopio del mundo proteico: puede identificar miles de proteínas diferentes en una muestra, medir cuántas hay de cada una, y detectar sus modificaciones. Es una tecnología probada, confiable, con enormes bases de datos de referencia. Pero tiene un talón de Aquiles que abre la puerta a nuevas tecnologías.
El problema fundamental es simple: la espectrometría de masas no puede ver moléculas individuales. Necesita aproximadamente mil millones de copias de la misma proteína para poder identificarla. Es como tratar de entender una conversación escuchando a un estadio entero hablando al mismo tiempo: solo escuchas el promedio, el ruido general, pero pierdes los detalles individuales.
Esto genera una cascada de limitaciones prácticas:
El problema de las proteínas raras: En una célula, algunas proteínas son superabundantes mientras otras existen en apenas unas copias. Es como tratar de escuchar un susurro en un concierto de rock. Las proteínas abundantes ahogan la señal de las raras, y son precisamente estas proteínas raras las que a menudo controlan si una célula se divide, muere, o se convierte en cancerosa. La diferencia de concentración puede ser de un billón de veces, pero los mejores instrumentos solo pueden detectar diferencias de mil o diez mil veces.
El problema de los gemelos idénticos: Algunos aminoácidos pesan exactamente lo mismo. La leucina y la isoleucina son como gemelos idénticos en una balanza: imposibles de distinguir por peso. Cuando trituras las proteínas en pedazos pequeños para analizarlas (que es lo que hace la espectrometría de masas), pierdes la información de cómo estaban organizados esos pedazos. Es como triturar un libro y tratar de reconstruir la historia pesando los fragmentos de papel.
El problema de la preparación: Antes de que puedas analizar una proteína, necesitas digerirla con enzimas, separarla, ionizarla (darle carga eléctrica), y a veces enriquecerla si tiene modificaciones especiales. Es un proceso que toma horas y donde cada paso puede salir mal. Algunas proteínas no se ionizan bien, otras se degradan en el proceso. Es como tener que cocinar cada muestra con una receta complicada antes de poder probarla, y a veces la cocción cambia el sabor original.
El problema del costo: Un espectrómetro de masas de alta gama cuesta entre $500K - $1M. Operarlo cuesta hasta $240 por hora. Necesitas un laboratorio especializado con control de temperatura, técnicos expertos, y software sofisticado. Es como si cada microscopio costara lo mismo que una casa y necesitara un piloto entrenado para operarlo. Esto significa que solo grandes instituciones pueden permitírselo, dejando fuera a laboratorios pequeños e investigadores independientes.
El mercado de espectrometría de masas continuará creciendo: MarketsandMarkets proyecta el sector de proteómica de $33.6B (2024) a $60.5B (2029) con CAGR del 12.4%, con la espectrometría manteniendo el 31.88% de participación tecnológica. Pero estas restricciones crean nichos distintos donde los enfoques alternativos ofrecen ventajas convincentes. La secuenciación de proteínas de molécula única aborda precisamente las aplicaciones donde falla el promediado de conjunto: variantes raras, discriminación de proteoformas, patrones de modificación postraduccional en moléculas individuales, y análisis de muestras preciosas donde no puedes permitirte consumir miles de millones de copias.
La Solución Semiconductora de Quantum-Si
Jonathan Rothberg es un emprendedor en serie con un historial impresionante. Ya había revolucionado la secuenciación de ADN dos veces: primero con 454 Life Sciences y luego con Ion Torrent, que convirtió máquinas del tamaño de refrigeradores en dispositivos de escritorio. Su trabajo contribuyó al Premio Nobel de Medicina de 2022. En 2013, se hizo la pregunta obvia: si los chips de computadora transformaron la lectura del ADN, ¿podrían hacer lo mismo con las proteínas?
La respuesta llegó en octubre de 2022, cuando su equipo publicó en Science el sistema Platinum de Quantum-Si: el primer dispositivo del mundo que puede leer proteínas una molécula a la vez usando tecnología de chips semiconductores.
Cómo funciona: La magia del chip
Imagina un chip de computadora del tamaño de una uña que contiene millones de pozos microscópicos. Cada pozo es tan pequeño que solo cabe una molécula de proteína. Es como tener millones de microscopios trabajando en paralelo, cada uno observando una sola molécula. Todo esto cabe en una máquina del tamaño de una impresora grande que puedes poner sobre tu escritorio.
El proceso es ingenioso en su simplicidad:
1. Preparar la muestra (3 horas) Primero cortas las proteínas en pedazos más pequeños llamados péptidos, como cortar un collar largo en segmentos manejables. Le agregas una especie de “manija química” a cada pedazo para poder manipularlo. Luego distribuyes estos péptidos en los millones de pozos del chip: un péptido por pozo.
2. El juego del reconocimiento Aquí viene la parte brillante. Imagina que tienes 20 tipos diferentes de llaves fluorescentes, cada una diseñada para reconocer uno de los 20 aminoácidos. Estas “llaves” (proteínas reconocedoras) fluyen sobre el chip. Cuando una llave encuentra su cerradura correspondiente (el aminoácido correcto), se pega temporalmente y brilla.
3. La firma del tiempo (la innovación clave) La mayoría pensaría: “OK, vemos qué color brilla y ya sabemos qué aminoácido es”. Pero Quantum-Si fue más allá. Miden cuánto tiempo se queda pegada cada llave, con qué intensidad brilla, y el patrón de cómo se pega y se suelta. Es como identificar a alguien no solo por su voz, sino por su ritmo al hablar, sus pausas, su entonación. Cada aminoácido tiene su propia “personalidad temporal” que además cambia según qué aminoácidos tenga como vecinos. Youtube hace esto para todos los videos con audio e imágenes genera un “fingerprint” de todo su contenido digital, es así como funciona el copyright system a escala.
4. Quitar y repetir Una enzima especial corta el primer aminoácido de cada péptido en todos los millones de pozos al mismo tiempo. Es como pelar capas de una cebolla de manera perfectamente sincronizada. Esto expone el siguiente aminoácido, y el ciclo se repite. Como usan enzimas naturales en lugar de químicos agresivos, el proceso es suave y confiable.
5. La inteligencia artificial entra en escena Un software llamado ProteoVue, entrenado con inteligencia artificial, interpreta todos estos patrones de tiempo y brillos. Actualmente puede identificar directamente 13 de los 20 aminoácidos, y usa el contexto para inferir los demás. Es como un detective que puede identificar a la mayoría de los sospechosos por su cara, y a los demás por su manera de caminar y comportarse.
Los números que importan
El sistema actual puede leer millones de moléculas en paralelo. Cada medición toma milisegundos. No necesita amplificar nada porque literalmente está viendo moléculas individuales. El nuevo modelo Platinum Pro (enero 2025) es más fácil de usar y puede procesar datos tanto en la nube como localmente.
Pero lo más interesante está por venir. La plataforma Proteus, que llegará a fines de 2026, promete leer miles de millones de moléculas por corrida. Es como pasar de leer un libro a la vez a leer una biblioteca entera. El objetivo final es la “secuenciación de novo”: leer proteínas completamente desconocidas sin necesidad de compararlas con una base de datos, algo que revolucionaría el descubrimiento de nuevas proteínas.
El negocio detrás de la ciencia
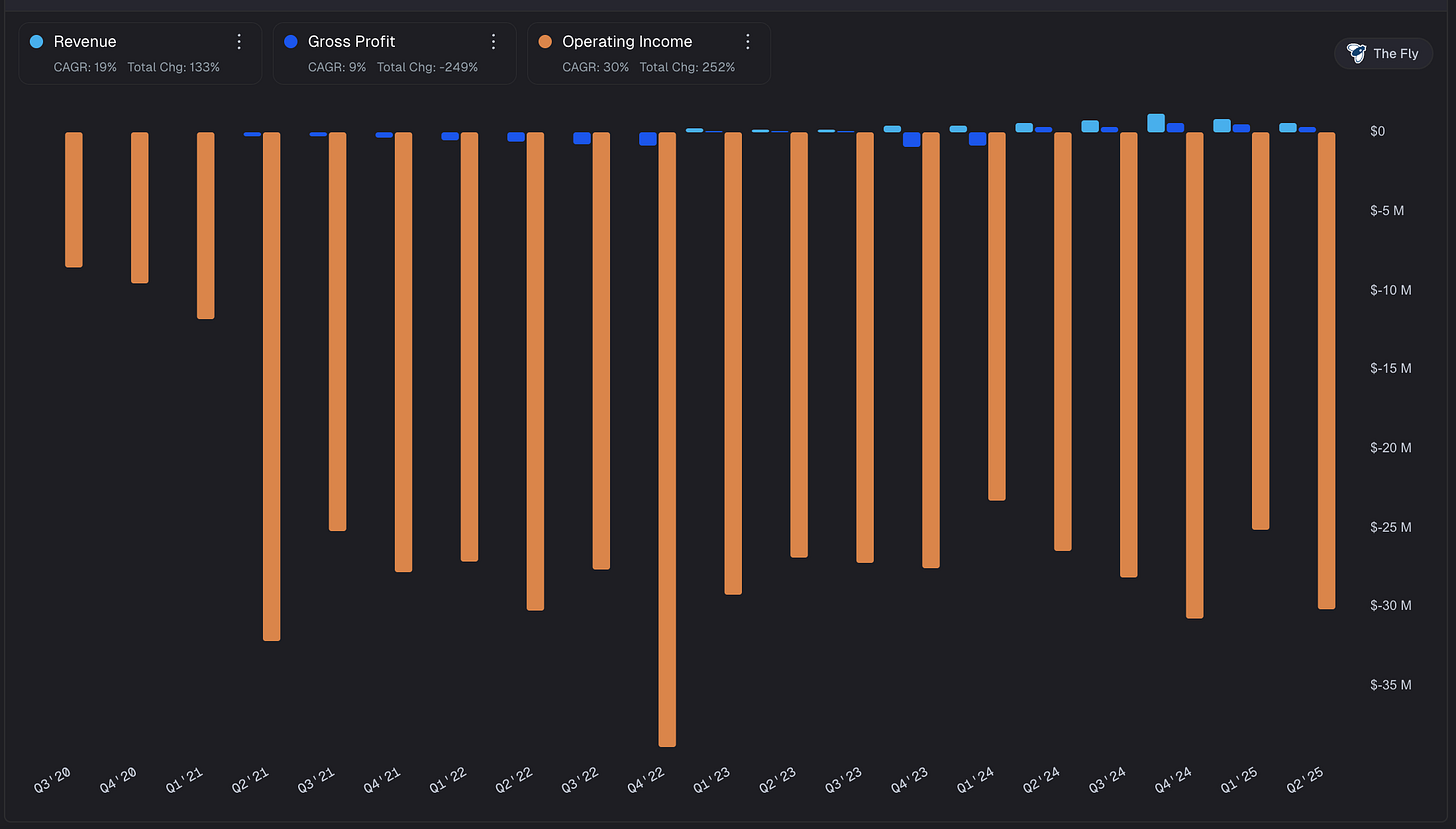
Los números financieros cuentan una historia de crecimiento temprano pero prometedor. Los ingresos casi se triplicaron (aun pequeños en escala) de 2023 a 2024 (de $1.08M a $3.1M). Todavía pierden dinero (-$101M en 2024), pero es normal para una empresa en esta etapa. Tienen $214 millones en el banco, suficiente para operar hasta mediados de 2028 sin necesitar más inversión.
Lo más impresionante es su fortaleza de patentes: más de 1,000 patentes que protegen todo, desde las moléculas reconocedoras hasta el diseño del chip y los algoritmos. Un moat de puro IP.
Validación Estratégica: NVIDIA y ARK Invest
El gigante de la AI entra en escena
El 20 de noviembre de 2024, Quantum-Si anunció algo que hizo que su acción subiera 119% en un solo día: una colaboración con NVIDIA. Aunque NVIDIA no invirtió dinero directamente (proporciona tecnología, no capital), el mensaje: la empresa más valiosa del mundo de AI cree que QSI tiene algo especial.
¿Por qué le importa a NVIDIA? Simple: datos. Cuando QSI secuencia proteínas, genera cantidades masivas de información que necesitan procesarse en tiempo real. Es como tratar de beber de una manguera de bomberos. George Vacek de NVIDIA lo explicó claramente: secuenciar proteínas requiere poder computacional extremo. Y cuando la plataforma Proteus llegue en 2026, pasarán de millones a miles de millones de lecturas por corrida. Sin la infraestructura de NVIDIA, sería como tratar de editar videos en 4K con una computadora de los 90s.
Cathie Wood apuesta
ARK Invest, el fondo liderado por Cathie Wood famoso por sus apuestas en tecnología disruptiva, tiene una posición significativa en QSI. Actualmente poseen entre 9-12% de todas las acciones de la empresa, convirtiéndolo en una de las 30 principales posiciones de su fondo de genómica (ARKG).
ARK entró temprano, participando en la ronda de inversión de $425 millones cuando QSI salió a bolsa en junio de 2021 via un SPAC. Han mantenido su posición a pesar de la volatilidad, aunque han tomado algunas ganancias recientemente (cosa normal cuando una inversión se ha apreciado).
La lógica de Cathie Wood. En octubre de 2024, ella explicó: “Estamos viendo la convergencia de secuenciación, inteligencia artificial y edición genética para curar enfermedades... Las semillas que se plantaron hace 25-30 años ahora están floreciendo”. Para ARK, la proteómica es el siguiente paso lógico después de la genómica.
Su argumento: mientras que tenemos solo 20,000 genes humanos, estos producen más de 220 millones de proteínas diferentes. Es como si los genes fueran el alfabeto y las proteínas fueran todos los libros que puedes escribir con esas letras. Y son las proteínas, no los genes, las que realmente hacen el trabajo en nuestro cuerpo. Cuando te enfermas, generalmente es porque algo anda mal con tus proteínas, no con tus genes.
La visión de ARK es que la convergencia de herramientas multiómicas (genómica, proteómica, metabolómica), inteligencia artificial y medicina de precisión está transformando la salud de una práctica basada en promedios poblacionales a una ciencia personalizada basada en datos individuales. Y las empresas que pueden leer proteínas individuales con chips semiconductores están en el centro de esta transformación.
Máquinas Moleculares: Por qué Importa la Sensibilidad de Molécula Única
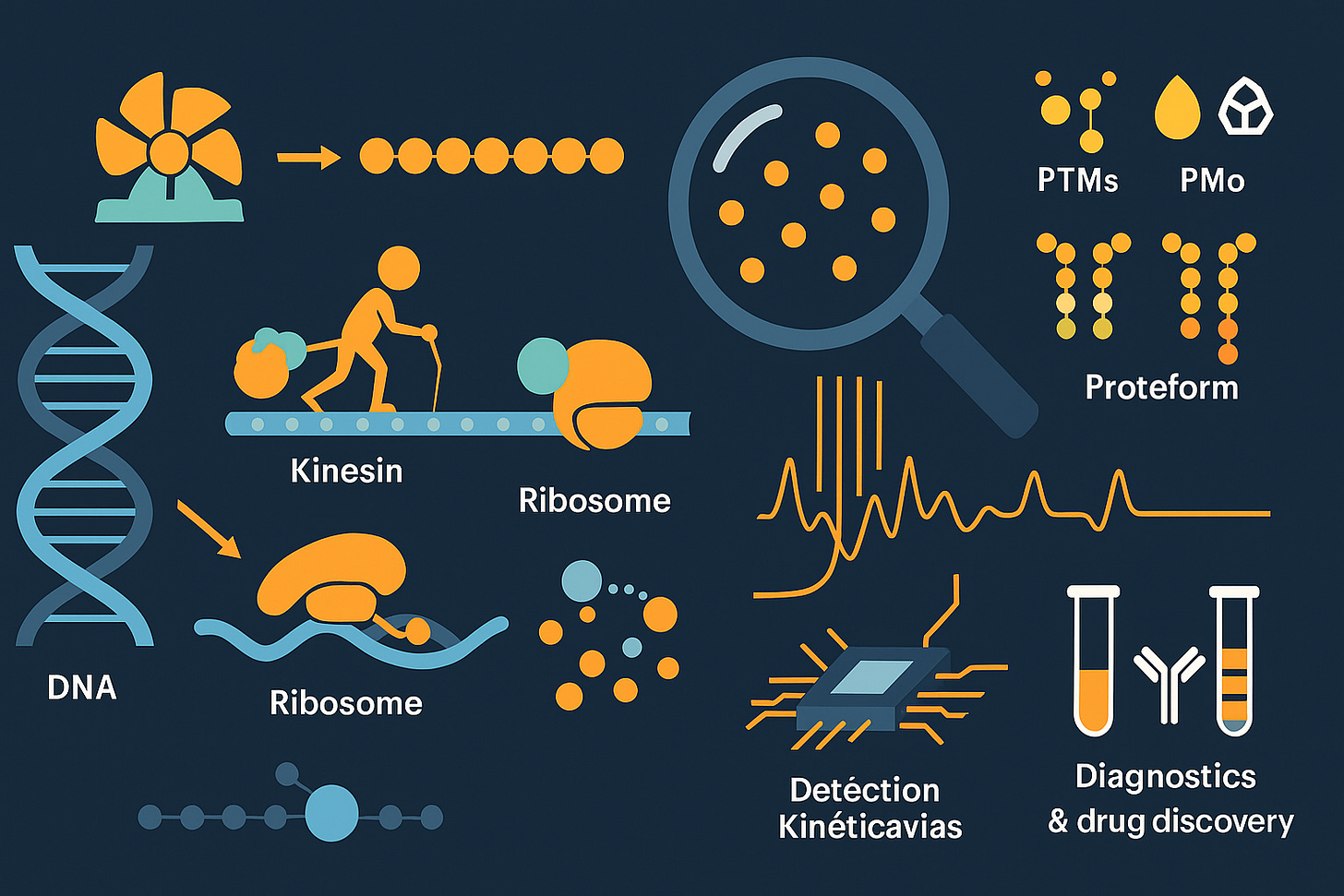
Las proteínas no son estructuras estáticas. Son máquinas moleculares: ensamblajes de componentes que producen movimientos mecánicos en respuesta a estímulos. La ATP sintasa aprovecha los gradientes de protones para hacer girar una turbina, sintetizando la moneda energética de las células. La miosina se desplaza a lo largo de los filamentos de actina, contrayendo los músculos. Las kinesinas caminan a lo largo de los microtúbulos, transportando carga. La ARN polimerasa descomprime el ADN y transcribe genes. El ribosoma une aminoácidos en proteínas, coordinando más de 600 proteínas en ensamblajes complejos.
Estas máquinas exhiben las características distintivas de los sistemas diseñados: modularidad, función cíclica, consumo de energía y movimiento coordinado de partes separadas. Representan los sistemas autónomos más pequeños capaces de sostener procesos dinámicos cuando se les suministra energía y sustratos. Y son ubicuas: prácticamente todos los procesos celulares dependen de máquinas moleculares.
Su relevancia clínica es directa. La disfunción en las máquinas moleculares causa enfermedades. Las proteínas mal plegadas se agregan en Alzheimer y Parkinson. Las quinasas mutadas impulsan el cáncer. Los trastornos autoinmunes surgen cuando las máquinas receptoras inmunes identifican erróneamente lo propio como extraño. Más del 95% de los objetivos farmacológicos son proteínas, no genes. Comprender cómo funcionan estas máquinas, y por qué fallan, requiere analizar sus estructuras, modificaciones y comportamiento dinámico.
Aquí es donde la sensibilidad de molécula única se vuelve esencial. Consideremos el desarrollo de anticuerpos. Un anticuerpo terapéutico podría diferir de un candidato fallido por una sola sustitución de aminoácido o por un patrón de glicosilación que afecta la vida media y la inmunogenicidad. El análisis masivo ve el promedio. La secuenciación de molécula única ve cada variante. Para mezclas de anticuerpos policlonales después de la vacunación, los métodos de molécula única pueden extraer secuencias individuales de anticuerpos de fondos complejos: QSI demostró esto para las respuestas a la vacuna COVID-19, logrando en dos semanas lo que los métodos tradicionales luchan por lograr en absoluto.
Las modificaciones postraduccionales a menudo ocurren subestequiométricamente: solo una fracción de las copias de proteínas llevan la modificación en cualquier momento. La fosforilación de una proteína de señalización podría alcanzar un máximo del 30% de ocupación, pero ese 30% determina si la célula prolifera o muere. Los métodos de conjunto informan el promedio; la detección de molécula única cuenta las poblaciones modificadas y no modificadas por separado. La capacidad de detectar PTMs en tiempo real a medida que procede la secuenciación—lo que permiten las firmas cinéticas de Quantum-Si—proporciona información inalcanzable a través de métodos que requieren pasos de enriquecimiento.
La discriminación de proteoformas importa clínicamente. Las isoformas de tropomiosina difieren solo por unos pocos aminoácidos pero tienen funciones distintas en el músculo. Las variantes de un solo aminoácido en proteínas asociadas al cáncer determinan la sensibilidad al fármaco. Quantum-Si demostró la discriminación de proteoformas de tropomiosina y publicó el análisis de interleucina-6 en colaboración con Northwestern, mostrando resolución de un solo aminoácido para regiones críticas para la unión al receptor, directamente relevante para la investigación terapéutica del cáncer y autoinmune.
Las capacidades revolucionarias desbloquean varias categorías de aplicación:
Descubrimiento de fármacos: Identificar anticuerpos terapéuticos, caracterizar variantes de secuencia que afectan la afinidad de unión, validar el desarrollo de biosimilares, descubrir nuevos inhibidores de puntos de control inmunológico. Liberate Bio usa el código de barras de proteínas de QSI para examinar vehículos de entrega de nanopartículas lipídicas para terapia génica, acelerando el desarrollo con información en tiempo real.
Diagnóstico: Descubrir biomarcadores en sangre, orina o líquido cefalorraquídeo para la detección temprana de enfermedades. La proteína PAK2 identificada como regulada al alza en el cáncer de pulmón de células no pequeñas ejemplifica biomarcadores potenciales. El monitoreo del rechazo de trasplante renal a través de proteómica urinaria ofrece precisión no invasiva imposible con genómica.
Medicina personalizada: Comparar proteomas de pacientes individuales con líneas base saludables para crear planes de tratamiento personalizados. Estratificar pacientes como respondedores versus no respondedores basándose en firmas de proteínas. Monitorear la eficacia terapéutica a través de cambios proteómicos con el tiempo.
Investigación básica: Caracterizar la estructura, función y dinámica de las proteínas con resolución de molécula única. Mapear interacciones proteína-proteína. Estudiar el tráfico de proteínas para dilucidar mecanismos de enfermedad. Facilitar la biología estructural proporcionando secuencias precisas para cristalografía y RMN.
La aplicación de código de barras de proteínas merece atención particular. Al codificar información en secuencias de aminoácidos—usando péptidos abióticos o diseñados como códigos de barras moleculares—los investigadores pueden multiplexar experimentos. La plataforma de QSI permite examinar 24 candidatos en un solo experimento agrupado con un rango dinámico de 120-240 veces. Esto acelera los experimentos de evolución dirigida, el cribado CRISPR y la selección de candidatos terapéuticos.
Oportunidad de Mercado y Posición Competitiva
El mercado de proteómica presenta una oportunidad sustancial con dinámicas de crecimiento razonablemente bien establecidas. Múltiples firmas de investigación proyectan el mercado de $27-36B (2024) a $44-145B para 2030-2034, con un CAGR de consenso alrededor del 12-15%. América del Norte domina con 44-47% de participación de mercado. Las aplicaciones de descubrimiento de fármacos representan el 47-52% de los ingresos. Los reactivos y consumibles representan el 70-72% del mercado, con los instrumentos comprendiendo el resto.
El panorama competitivo se estratifica en 3 niveles:
Nivel 1: Incumbentes de espectrometría de masas—Thermo Fisher (plataformas Orbitrap, adquisición de Olink), Danaher (Sciex), Agilent, Waters, Bruker. Estas empresas dominan la mayor parte del mercado de proteómica con tecnología madura y validada. No son objetivos de desplazamiento; la espectrometría de masas seguirá siendo el caballo de batalla para el análisis masivo de alto rendimiento. Pero sus restricciones—requisitos de sensibilidad, carga de infraestructura, ambigüedad de isómeros—crean nichos.
Nivel 2: Plataformas proteómicas de próxima generación—Nautilus Biotechnology (plataforma basada en afinidad con anticuerpos de epítopo corto, afirma 95% de cobertura del proteoma), SomaLogic (basado en aptámeros), Olink (ensayo de extensión de proximidad), Quanterix (Simoa). Estas empresas atacan varios puntos problemáticos: profundidad de cobertura, multiplexación, automatización. Nautilus recaudó $514M a través de SPAC y tiene $374M en efectivo, posicionándolos como el competidor más cercano de QSI en la categoría de secuenciación de molécula única. Sin embargo, Nautilus sigue siendo pre-comercial con asociaciones farmacéuticas en lugar de ventas de productos. Su actual market cap $160M
Nivel 3: Secuenciación emergente de molécula única—Quantum-Si se mantiene esencialmente solo como un secuenciador comercialmente disponible de proteínas de molécula única. La secuenciación de proteínas basada en nanoporos permanece en fases de investigación académica. Oxford Nanopore Technologies (exitoso en secuenciación de ADN) ha expresado interés pero enfrenta desafíos físicos sustanciales controlando la translocación de proteínas plegadas y con carga neutra a través de nanoporos.
El posicionamiento competitivo de Quantum-Si enfatiza varios diferenciadores:
Ventaja del primer jugador: Producto comercialmente disponible con validación revisada por pares (Science, 2022) e instalaciones de clientes generando ingresos. Nautilus apunta al lanzamiento comercial pero actualmente opera en modo de asociación.
Escalabilidad semiconductora: La fabricación a través de SkyWater Technology aprovecha la cadena de suministro semiconductora de un billón de dólares. Se aplica la economía de la Ley de Moore: a medida que mejora la fabricación de chips, los costos disminuyen y las capacidades aumentan. Esto contrasta con la fabricación de reactivos biológicos (anticuerpos de Nautilus) o la fabricación de instrumentos físicos (espectrometría de masas).
Accesibilidad: Precio de $50K versus $100K-$1M para espectrometría de masas. Uso de escritorio versus instalaciones dedicadas. Requisitos mínimos de experiencia versus operadores especialistas. Estos factores expanden el mercado direccionable más allá de las instalaciones centrales a laboratorios individuales.
Ventajas técnicas: Sensibilidad de molécula única, contenido de información cinética, detección directa de PTM, discriminación de proteoformas, flujos de trabajo simplificados. Estos crean nichos defendibles incluso cuando mejora la espectrometría de masas.
Fundador probado: El historial de Rothberg (454 Life Sciences revolucionó la secuenciación de ADN, Ion Torrent la llevó a uso de escritorio) proporciona credibilidad. Su filosofía de “democratización”—hacer que las herramientas analíticas poderosas sean accesibles para cada laboratorio—se alinea con las estrategias de expansión del mercado.
Asociaciones estratégicas: NVIDIA (AI/computación), Avantor (distribución en América del Norte), SkyWater (fabricación), Planet Innovation (desarrollo de Proteus). Estas asociaciones reducen el riesgo de ejecución en frentes críticos.
Qué me Gusta de QSI 0.00%↑
Lo que me da confianza:
La tecnología funciona: No es humo. Tienen un producto real, publicado en Science, ganando premios de la industria, con clientes usando la máquina y generando datos publicables. Los ingresos están creciendo (se triplicaron de 2023 a 2024), lo que significa que alguien está comprando.
El problema es real: Las limitaciones de la espectrometría de masas no son inventadas. Realmente no puede ver moléculas individuales, realmente no puede distinguir ciertos aminoácidos, realmente cuesta una fortuna. El mercado de proteómica está creciendo al 12-15% anual, impulsado por tendencias sólidas como medicina personalizada y desarrollo de medicamentos biológicos.
Validación externa: Cuando NVIDIA colabora y Cathie Wood mantiene el 10% de tu empresa, algo están haciendo bien. Los primeros clientes (universidades y farmacéuticas) están adoptando la tecnología, lo que muestra que hay demanda real.
Escalabilidad creíble: El enfoque de semiconductores tiene sentido. Si funcionó para secuenciar ADN (Ion Torrent, Illumina), puede funcionar para proteínas. La plataforma Proteus prometida para 2026 no es ciencia ficción; es una evolución lógica de la tecnología actual.
El fundador tiene credibilidad: Rothberg no es un novato. Ya revolucionó la secuenciación de ADN dos veces. Sabe cómo llevar tecnología del laboratorio al mercado.

Riesgos reales:
Están quemando efectivo: Perdieron $101 millones en 2024 con solo $3 millones en ingresos. Eso es perder $33 por cada dólar que ganan. Tienen efectivo hasta 2028, pero si algo sale mal, necesitarán más dinero, lo que diluirá a los accionistas actuales.
La tecnología no está completa: Solo pueden identificar directamente 13 de 20 aminoácidos. Es como tener un alfabeto donde solo puedes leer claramente la mitad de las letras. La verdadera secuenciación “de novo” (leer proteínas completamente desconocidas) sigue siendo un sueño.
La competencia es feroz: Nautilus Biotechnology tiene $374 millones en el banco y dice tener mejor tecnología. Illumina, el gorila de $47B en genómica, acaba de entrar en proteómica. La espectrometría de masas no se va a quedar quieta. La ventana para establecerse puede cerrarse rápido.
La adopción será lenta: Los laboratorios no cambian de tecnología fácilmente. Tienen flujos de trabajo establecidos, personal entrenado, bases de datos construidas. Convencerlos de cambiar es como pedirle a alguien que cambie de iPhone a Android: técnicamente posible, prácticamente doloroso.
La valoración es especulativa: Con una capitalización de mercado de ~$300-400 millones y solo $3 millones en ingresos, estoy pagando futuro no presente. La acción ha caído más del 80% desde sus máximos. Es volátil (beta de 3.09 significa que se mueve tres veces más que el mercado).
El camino a la rentabilidad es largo: Con márgenes brutos del 58%, necesitarían ingresos de $150-200 millones solo para cubrir gastos. Eso es 50-70 veces los ingresos actuales. Estamos hablando de años, tal vez una década, antes de ver ganancias.
Es dificil de entender: Este substack lleva en modo borrador 3 semanas por que todo es nuevo: términos, conceptos, aplicaciones, tech, etc. No obstante se que con AI los avances en biología serán mas impresionantes que un agente que responde preguntas y por eso siempre he querido tener una punta en este mundo.
Síntesis: Por qué esto importa
Volvamos al principio. La paradoja de Levinthal nos mostró que las proteínas enfrentan un problema aparentemente imposible: tienen trillones de formas posibles pero encuentran la correcta en milisegundos. La naturaleza resolvió este rompecabezas mediante evolución, creando “embudos energéticos” que guían a las proteínas hacia su forma correcta. Pero nosotros, tratando de leer y entender estas proteínas, enfrentamos el mismo problema desde el otro lado.
La espectrometría de masas, nuestra herramienta actual, es como tratar de entender una conversación escuchando a un millón de personas hablar al mismo tiempo. Funciona bien cuando todas dicen más o menos lo mismo, pero pierde los susurros importantes, las voces únicas, los detalles que pueden marcar la diferencia entre un medicamento que funciona y uno que no.
Quantum-Si propone algo radicalmente diferente: escuchar una voz a la vez. Ver moléculas individuales. Contar cada proteína, medir su comportamiento único, capturar información que los métodos actuales simplemente no pueden ver. Y hacerlo con chips semiconductores que se vuelven más baratos y potentes cada año, siguiendo la misma curva que transformó las computadoras de cuartos enteros a dispositivos de bolsillo.
Los números son escalas que solo podemos comprender haciendo uso de AI. De 20,000 genes humanos surgen más de 220 millones de proteínas diferentes. Es la diferencia entre el alfabeto y toda la literatura escrita con esas letras. Los genes son el potencial; las proteínas son la acción. Y cuando algo sale mal en nuestro cuerpo, casi siempre es a nivel de proteínas, no de genes.
Quantum-Si representa una apuesta en varias transiciones simultáneas:
De leer genes a leer proteínas
De ver promedios a ver moléculas individuales
De máquinas del tamaño de refrigeradores a dispositivos de escritorio
De tecnología para pocos a herramientas para todos
¿Es arriesgado? Absolutamente. La tecnología está en pañales. La competencia es feroz. La adopción será lenta. Pueden pasar años antes de ver ganancias, si es que alguna vez las hay.
Pero aquí está el punto: los problemas difíciles crean las mayores oportunidades. La secuenciación de ADN parecía imposible en los 80s; ahora es rutina. Las proteínas son el siguiente frontera. Son más complejas, más dinámicas, más relevantes para la enfermedad. Quien resuelva cómo leerlas fácil y económicamente transformará la medicina.
La paradoja de Levinthal sigue sin resolverse completamente. AlphaFold puede predecir estructuras pero no explicar el proceso. Seguimos sin entender completamente cómo las proteínas hacen su magia. Pero por primera vez, tenemos herramientas que pueden ver esa magia ocurriendo, molécula por molécula, en tiempo real.
El 95% de los objetivos de medicamentos son proteínas. La mayoría de las enfermedades involucran proteínas mal plegadas o modificadas. La medicina personalizada requiere entender las proteínas de cada individuo.
El problema de las proteínas tiene que resolverse. La pregunta no es si, sino cuándo y quién. La combinación de semiconductores, AI y biología molecular que representa Quantum-Si tiene una oportunidad real de ser parte de esa solución. Es una apuesta a largo plazo en una categoría que está naciendo.
Obviamente todo lo anterior es NFA, ademas de acciones tengo varios lotes de buy calls al 2027







Gracias Hernán por este articulo tan interesante en un tema tan dificil de entender. Aún así y tomando datos de la Dieta de Información que he venido consumiendo, hace varios meses realice una compra pequena de acciones de QSI. Basicamente lo hice basado en lo que ustedes llaman "first principles" : Una producto/servicio único o dificilmente replicable, el equipo de trabajo, en especial el CEO, tiene condiciones de lider y todo el bagaje de conocimiento para creer que ve luz al final del tunel, alianzas claves que cimentan su credibilidad, entre otras razones. Lo expuesto por ti confirma que esta puede ser una muy buena opción a futuro, si se tiene paciencia !!
Hernán, muchas gracias por este tipo de contenido. Tremendo artículo!! Me volaste la cabeza con el tema de la proteómica. Esta vaina el realmente lo que moviliza el cambio de paradigma de la atención en salud. Y sumale a esto lo que hablaban con Antonio Linares en el capítulo de HIMS sobre las posibilidades que tiene una empresa como esta siendo top of the funnel de aprovecharse de todos estos nuevos péptidos abióticos que pueden empezar a salir con esta tecnología pa generar verticales de forma exponencial... una barbaridad pero que desde la ignorancia ya se puede percibir como algo tangible. Pa seguir aprendiendo y estudiando xq es muy complejo, pero hay muchísimo alpha!!